Produire un faisceau laser de rayons X ultra puissant pour visualiser une seule particule virale en un seul flash de quelques femtosecondes (10-15 seconde) : c’est la prouesse que vient de réaliser un consortium interdisciplinaire international de plus de 20 laboratoires (1), dont le laboratoire CNRS Information génomique et structurale. Ces chercheurs ont en effet reconverti l’accélérateur de particules de Stanford (SLAC) en un gigantesque appareil de radiologie pour particules « uniques » : cellules entières, virus, ou même macromolécules. Ces travaux publiés le 3 février 2011 dans la revue Nature annoncent une nouvelle ère pour la biologie structurale, en ouvrant l’utilisation des rayons X à l’étude de la structure tridimensionnelle d’objets biologiques asymétriques, non cristallisables, et même en mouvement. Les chercheurs travaillent désormais à améliorer la résolution des images pour parvenir à la visualisation détaillée de l’extérieur comme de l’intérieur de ces particules biologiques.
Les physiciens impliqués dans ces travaux étudient depuis le milieu des années 1990 la possibilité d’utiliser les accélérateurs de particules pour des études biologiques. Ils sont parvenus à adapter l’Accélérateur linéaire de Stanford (SLAC) en lui ajoutant toute une structure, composée d’aimants sur 800 mètres de long, pour produire, à partir de l’accélération des électrons, l’émission d’une quantité colossale de photons à la même fréquence dans les longueurs d’onde des rayons X durs. Ces photons constituent ainsi le faisceau laser de rayons X le plus puissant du monde. La puissance délivrée par cet instrument, le LCLS (Linac Coherent Light Sources), est de 6,5 1015 watts/cm2, soit une augmentation d’un facteur 10 milliards par rapport aux brillances disponibles jusqu’à présent ! Ce chiffre est difficilement concevable, quand on sait qu’un réacteur de centrale nucléaire délivre une puissance de 1000 mégawatts (1012 watts)… De plus, cette impulsion laser est très brève : 70 femtosecondes. Toute l’énergie envoyée sur la cible est ainsi concentrée en un temps extrêmement court, ce qui permet d’obtenir une image avant l’explosion de l’échantillon. Chaque particule biologique injectée dans le faisceau de photons, à une vitesse de 300km/h, est transformée en un plasma à une température de 100 000 degrés Kelvin. Mais auparavant, elle a eu le temps de diffracter 1,7 millions de photons à partir desquels est recréée son image, grâce à des méthodes mathématiques et des logiciels développés par les chercheurs.
Les aspects biologiques de ces travaux ont été assurés par l’équipe de Chantal Abergel dans le laboratoire CNRS Information génomique et structurale dirigé par Jean-Michel Claverie (professeur à l’Université de la Méditerranée). Les physiciens souhaitaient en effet valider ces travaux sur un objet biologique aussi « spectaculaire » que leur laser. Les chercheurs marseillais ont donc fourni l’objet d’étude, la particule de Mimivirus (2), et assuré la préparation et l’optimisation des échantillons nécessaires à l’expérience.
Ces travaux valident la faisabilité de l’utilisation d’un accélérateur de particules comme outil de biologie structurale pour obtenir en un seul « flash » laser la photographie de particules uniques (virus, bactéries, protéines, cellules…). Ils constituent une avancée « historique » dans le domaine de la biologie structurale. En effet, jusqu’à présent, deux techniques permettaient d’étudier des objets biologiques : la radiocristallographie et la microscopie électronique. Or les exigences requises par ces techniques (être « cristallisables », symétriques, statiques, et d’une taille adéquate) (3) excluent la plupart des objets biologiques et faussent souvent les résultats car elles supposent que toutes les molécules sont identiques et imposent une symétrie qui n’est souvent pas réelle. Avec le laser LCLS, toutes les particules pourront être étudiées individuellement, quelle que soit leur taille et leurs propriétés. Cette technique annonce ainsi une nouvelle ère : celle de la biologie structurale des objets uniques. L’Europe se prépare à relever ce nouveau défi avec son propre instrument, baptisé ELI, dont la mise en chantier vient de commencer (4).
Le laser LCLS va permettre d’étudier la surface mais aussi l’intérieur des particules puisque les rayons X traversent les échantillons. La résolution actuelle des images obtenues est de quelques nanomètres (1nm=10-9m). Les chercheurs travaillent actuellement pour optimiser ces performances et obtenir une résolution de l’ordre de quelques angströms (1Å=10-10m). Les expériences planifiées pour les mois prochains devraient permettre d’obtenir la structure tridimensionnelle complète (capside et nucléocapside interne) d’une particule de Mimivirus à l’échelle du nanomètre et de comparer différentes particules de Mimivirus entre elles. Les chercheurs pourront ainsi explorer pour la première fois l’existence d’un polymorphisme structural des particules virales.
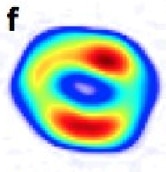
© Seibert et al.
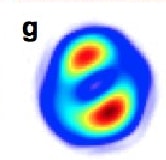
Reconstitution de l'image des deux particules virales de Mimivirus obtenue grâce au laser LCLS. © Seibert et al.
Notes :
(1) Ce consortium est dirigé par Janos Hajdu de l’Université d’Uppsala en Suède.
(2) Découvert en 2003 par les équipes CNRS de Didier Raoult et de Jean-Michel Claverie à Marseille, Mimivirus est le plus gros virus à ADN connu à ce jour.
(3) La radiocristallographie utilise la cristallisation d’un grand nombre d’objets identiques selon un réseau régulier pour amplifier leur signal individuel de diffraction. La microscopie électronique utilise les symétries (réelles ou approximative) des objets pour en reconstituer la structure tridimensionnelle à partir d’une multitude (des dizaines de milliers) d’images de leur projection sur un plan.
(4) Voir le communiqué de presse CNRS : Consulter le site web
Références :
Single Mimivirus particles intercepted and imaged with an X-ray laser.
M. Marvin Seibert, Tomas Ekeberg, Filipe R. N. C. Maia, Martin Svenda, Jakob Andreasson, Olof Jönsson, Duško Odi?, Bianca Iwan, Andrea Rocker, Daniel Westphal, Daniel P. DePonte, Anton Barty, Joachim Schulz, Lars Gumprecht, Nicola Coppola, Andrew Aquila, Mengning Liang, Thomas A. White, Andrew Martin, Carl Caleman, Stephan Stern, Chantal Abergel, Virginie Seltzer, Jean-Michel Claverie, Christoph Bostedt, John D. Bozek, Sébastien Boutet, A. Alan Miahnahri, Marc Messerschmidt, Jacek Krzywinski, Garth Williams, Keith O. Hodgson, Michael J. Bogan, Christina Y. Hampton, Raymond G. Sierra, Dmitri Starodub, Inger Andersson, Saša Bajt, Miriam Barthelmess, John C. H. Spence, Petra Fromme, Uwe Weierstall, Richard Kirian, Mark Hunter, R. Bruce Doak, Stefano Marchesini, Stefan P. Hau-Riege, Matthias Frank, Robert L. Shoeman, Lukas Lomb, Sascha W. Epp, Robert Hartmann, Daniel Rolles, Artem Rudenko, Carlo Schmidt, Lutz Foucar, Nils Kimmel, Peter Holl, Benedikt Rudek, Benjamin Erk, André Hömke, Christian Reich, Daniel Pietschner, Georg Weidenspointner, Lothar Strüder, Günter Hauser, Hubert Gorke, Joachim Ullrich, Ilme Schlichting, Sven Herrmann, Gerhard Schaller, Florian Schopper, Heike Soltau, Kai-Uwe Kühnel, Robert Andritschke, Claus Dieter Schröter, Faton Krasniqi, Mario Bott, Sebastian Schorb, Daniela Rupp, Marcus Adolph, Tais Gorkhover, Helmut Hirsemann, Guillaume Potdevin, Heinz Graafsma, Björn Nilsson, Henry N. Chapman, Janos Hajdu.
Nature, 3 février 2011
Source: communiqué de presse du CNRS
Laisser un commentaire