Une équipe internationale de scientifiques, dont Thierry Wirth et Raphaël Leblois de l’unité « Origine, structure et évolution de la biodiversité » (Muséum national d’Histoire naturelle/CNRS/EPHE) viennent de détailler pour la première fois l’histoire évolutive de la peste grâce aux avancées liées aux nouvelles technologies de séquençage et de typage génétique. Les informations cumulées à partir de 17 génomes complets et une collection unique de près de 300 souches, ont permis aux scientifiques de reconstituer l’origine biogéographique de cette maladie, son âge et sa dispersion en vagues épidémiques successives. Les résultats de cette étude sont publiés en ligne sur le site du magazine Nature Genetics le 31 octobre.
La peste est l’un des fléaux les plus importants de notre civilisation. Le plus souvent mortelle quand elle est non traitée, cette maladie a profondément affecté nos sociétés. Son agent infectieux est une bactérie en forme de bâtonnet, Yersinia pestis, et son réservoir naturel est essentiellement composé de diverses populations et espèces de rongeurs. La transmission entre hôtes se fait par des piqûres de puces.
Des reconstructions phylogénétiques ont permis de montrer que les souches chinoises sont les plus ancestrales et qu’elles présentent la plus forte diversité génétique. Ces résultats impliquent que la peste serait apparue en Chine il y a plus de 2600 ans. De cette région elle a ensuite essaimé vers l’Europe occidentale le long de la route de la soie (débutée il y a plus de 600 ans), vers l’Afrique (sans doute importée sur ce continent par le navigateur Zheng He au XVe siècle) et plus tardivement vers l’Amérique du Nord et Madagascar, à la fin du XIXe siècle.
Les données moléculaires ont permis de retracer avec une rare précision l’arrivée de la peste aux Etats-Unis via la Chine et Hawaii et la propagation du germe depuis les ports de San Francisco et Los Angeles vers l’intérieur des terres. Ces travaux ont également permis de déterminer des lignées évolutives inféodées à certaines régions géographiques qui se distinguent par des mutations spécifiques ; mais ils ont surtout démontré que l’ensemble des vagues épidémiques successives ont eu pour source l’Asie centrale et la Chine.
Cette étude servira sans aucun doute de paradigme pour l’étude d’autres bactéries hautement pathogènes et peu variables génétiquement, tels les agents infectieux de la tuberculose ou de l’anthrax. Elle démontre également l’utilité des approches évolutives dans notre compréhension des maladies émergentes et autres infections bactériennes.
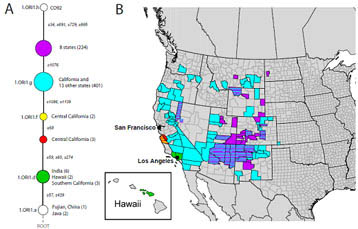
Vague de propagation de la peste aux Etats-Unis reconstituée à partir des polymorphismes nucléotidiques des souches américaines. Noter la progression vers l'ouest qui s'accompagne de nouvelles mutations. @CNRS
Références :
Giovanna Morelli, Yajun Song, Camila J. Mazzoni, Mark Eppinger,, Philippe Roumagnac, David M. Wagner, Mirjam Feldkamp, Barica Kusecek, Amy J. Vogler, Yanjun Li, Yujun Cui, Nicholas R. Thomson, Thibaut Jombart, Raphael Leblois, Peter Lichtner, Lila Rahalison, Jeannine M. Petersen, Francois Balloux, Paul Keim, Thierry Wirth, Jacques Ravel, Ruifu Yang, Elisabeth Carniel & Mark Achtman. Phylogenetic diversity and historical patterns of pandemic spread of Yersinia pestis. Nature Genetics, published online 31 october 2010. DOI : 10.1038/ng.705.
Source: communiqué de presse du CNRS
Laisser un commentaire