Un an après l’attribution du prix Nobel de chimie pour la découverte de la structure atomique du ribosome bactérien, les chercheurs de l’Institut de génétique et de biologie moléculaire et cellulaire (CNRS/Université de Strasbourg/Inserm) viennent de déterminer la première structure d’un ribosome eucaryote, celui de la levure. Ces travaux publiés le 26 novembre 2010 dans la revue Science mettent fin à une course internationale effrénée pour la détermination de la structure de cette imposante machinerie cellulaire. Le ribosome eucaryote est actuellement la plus grande molécule asymétrique biologique dont la structure a été élucidée par cristallographie. Ces résultats ouvrent de nouvelles pistes de recherche pour la compréhension de la dynamique de la synthèse protéique et pour le développement de nouveaux composés thérapeutiques.
Le ribosome, une « nanomachine » complexe
Le ribosome est une machine essentielle de la cellule qui assure la synthèse protéique à partir de l’information génétique. Il interagit avec de nombreuses protéines et occupe un rôle clé dans divers processus cellulaires. Depuis longtemps, les chercheurs tentent de déterminer sa structure atomique, défi considérable au vu de sa taille et de sa complexité. Le ribosome bactérien a une structure semblable mais non identique à celle du ribosome eucaryote (non bactérien). Il est plus petit (seulement 2.3 MDa(1) contre 3,3MDa pour le ribosome eucaryote) mais présente la même organisation générale en deux sous-unités. En 2009, le prix Nobel de chimie récompensait les chercheurs qui avaient déterminé pour la première fois la structure du ribosome bactérien. Celle de son homologue eucaryote faisait depuis l’objet d’une course effrénée.
La structure du ribosome eucaryote, une détermination difficile
Pour déterminer la structure du ribosome eucaryote, les chercheurs se sont intéressés à celui de la levure, un organisme modèle idéal, déjà connu et largement utilisé en biologie. Avec une masse d’environ 3.3 MDa, le ribosome eucaryote est plus gros de 40 pour cent par rapport à son homologue bactérien. Après de longs travaux de purification et de stabilisation de la molécule, les chercheurs strasbourgeois ont finalement obtenu sa structure atomique avec une très bonne résolution (de 0,415 nanomètres, soit une résolution à l’échelle de la molécule). L’équipe de l’Institut de génétique et de biologie moléculaire et cellulaire a confirmé l’existence de mouvements au sein des sous-unités du ribosome, mais également l’une par rapport à l’autre, mettant en évidence la dynamique de pivotement à l’origine du mécanisme de la synthèse protéique.
Des résultats prometteurs
Prochains objectifs pour l’équipe : déterminer la structure du ribosome d’autres eucaryotes mais également améliorer encore la résolution des résultats pour obtenir une description du ribosome et des mécanismes qui s’y déroulent à l’échelle atomique. La connaissance de cette structure facilitera la compréhension des relations structure/fonction à l’échelle atomique et fournira les bases moléculaires pour l’investigation des caractéristiques uniques de la machinerie traductionnelle des eucaryotes. Une telle description apportera également de précieuses informations pour le développement de nouveaux composés thérapeutiques ciblant les virus, les protozoaires (paludisme, maladie du sommeil, toxoplasmose, etc.), les champignons et les bactéries. En effet, en bloquant le ribosome de ces organismes, on en bloquerait toute activité.
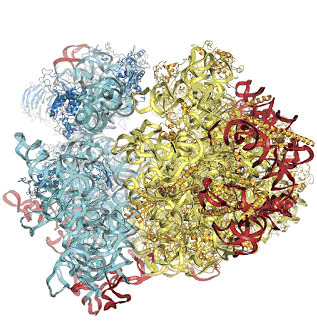
Vue de la structure du ribosome de levure : la petite sous-unité est représentée en bleu tandis que la grande apparaît en jaune. L'ARN ribosomique est représenté en rouge. © M. Yusupov
Notes :
(1) Unité de masse souvent utilisée par les biochimistes, le Dalton, qui est la masse d’un atome d’hydrogène, vaut 1,67.10-24 g. Il est symbolisé par Da. MDa signifie megaDalton soit un millions de Dalton.
Références :
Crystal structure of the eukaryotic ribosome. Adam Ben-Shem, Lasse Jenner, Gulnara Yusupova and Marat Yusupov. Science, 26 novembre 2010.
Source: communiqué de presse du CNRS
Laisser un commentaire