Des chercheurs du CEA (1), du CNRS, de l’Inserm, de l’Inra et de l’Université Joseph Fourier ont élaboré AT_Chloro, une base de données protéomiques unique dédiée au chloroplaste (2) de la plante modèle Arabidopsis thaliana, accessible sur internet. La base AT_Chloro n’est pas seulement un répertoire de protéines, puisqu’elle va au-delà en présentant des données semi-quantitatives qui permettent de déterminer dans quel sous compartiment du chloroplaste se trouvent les protéines identifiées. Grâce aux données d’une grande précision qu’elle contient, cette base constitue un outil des plus intéressants pour la communauté scientifique puisqu’elle ouvre aussi la voie à l’analyse beaucoup plus rapide de la dynamique du protéome chloroplastique. A la clé, des applications dans des domaines aux enjeux aussi variés que la qualité de la biomasse, l’impact des perturbations de l’environnement, la culture en milieux carencés… La base AT_Chloro peut désormais être interrogée par les chercheurs du monde entier sur Consulter le site web .
L’essentiel du carbone renouvelable est fixé par les organismes photosynthétiques grâce à leurs chloroplastes, siège de la photosynthèse. En outre, les chloroplastes réalisent de nombreuses fonctions essentielles comme la synthèse d’acides gras, de lipides ou d’acides aminés, qui nécessitent le fonctionnement coordonné de trois sous-compartiments : les thylacoïdes, le stroma et l’enveloppe (voir schéma).
L’approfondissement des connaissances des voies métaboliques du chloroplaste et de leur régulation est déterminant dans la perspective d’une meilleure valorisation de la biomasse végétale. C’est également essentiel si l’on veut analyser l’impact des variations de l’environnement sur les plantes.
Grâce à une stratégie d’analyse protéomique mise en œuvre pour la première fois chez Arabidopsis thaliana les chercheurs ont identifié un grand nombre de protéines du chloroplaste, déterminé leur localisation dans ses différents sous-compartiments et construit la base de données AT_Chloro. Cette dernière a ainsi été établie en réalisant près de 500 analyses sur des fractions hautement purifiées des trois sous compartiments majeurs du chloroplaste. Des informations précises ont été obtenues pour 10 654 peptides distincts correspondant à 1 323 protéines validées. Un des enjeux actuels de la protéomique est la réalisation d’études comparatives afin de suivre des processus dynamiques. Dans cette optique, la base AT_Chloro et l’utilisation de la méthode quantitative AMT (3), permet, à partir d’une seule et même analyse, d’identifier et de quantifier les protéines du chloroplaste.
Les chercheurs ont également poussé plus loin l’analyse protéomique et déterminé la localisation des protéines de la base dans les sous compartiments en utilisant la méthode dite de Spectral Count (4). Ils ont caractérisé en particulier les protéines de l’enveloppe du chloroplaste. Ainsi, près de 500 protéines (sur les 1 323 protéines identifiées) sont en fait situées dans l’enveloppe alors qu’elles ne représentent en masse que 1 à 2% des protéines du chloroplaste.
AT_Chloro est unique par la précision de ses données expérimentales (masse exacte des peptides, temps de rétention dans le spectromètre de masse) qui vont donner aux chercheurs la possibilité de réaliser des analyses des protéomes chloroplastiques en utilisant des technologies moins complexes et moins consommatrices de temps et d’argent. De plus, elle offre désormais à la communauté scientifique la possibilité d’effectuer des requêtes pour initier des analyses quantitatives sur des protéines présentes dans les divers compartiments des chloroplastes d’Arabidopsis thaliana. Son utilisation permettra des avancées dans des domaines aussi variés que l’adaptation des plantes aux perturbations de leur environnement (contamination par des métaux lourds, cultures en milieux carencés, variations de température ou de teneur en CO2), ou l’amélioration de la biomasse végétale pour des applications dans les domaines de l’énergie ou de la chimie verte.
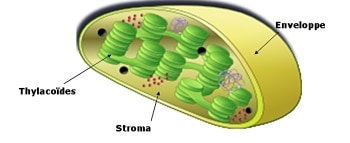
représentation schématique de l'intérieur d'un chloroplaste. Il contient trois compartiments principaux. Le système membranaire interne (thylacoïdes, en vert) est essentiellement le siège de la phase lumineuse de la photosynthèse. Ces thylacoïdes baignent dans un milieu intra-chloroplastique (le stroma) qui est le siège de nombreux métabolismes essentiels, en particulier la fixation du CO2 (phase dite sombre de la photosynthèse). L'enveloppe (la double membrane qui délimite l'organite, de couleur jaune) contrôle principalement les échanges entre le chloroplaste et les autres compartiments de la cellule végétale © Copyright EDyP/CEA
Accès à la base AT_Chloro http://www.grenoble.prabi.fr/protehome/grenoble-plant-proteomics/
Notes :
(1) Laboratoire d’Etude de la dynamique des protéomes (CEA – Inserm U880 – UJF) et Laboratoire de Physiologie cellulaire végétale (UMR CNRS 5168 – CEA – Inra – UJF) de l’iRTSV, Institut de recherches en technologies et sciences pour le vivant, Direction des sciences du vivant, Grenoble.
(2) Le chloroplaste est une structure cellulaire, siège de la photosynthèse chez les végétaux.
(3) La méthode AMT (Accurate Mass and time Tags) repose sur l’utilisation de la spectrométrie de masse à transformée de Fourier, qui permet de déterminer avec une très grande précision la masse des peptides provenant des protéines après digestion par la trypsine.
(4) La méthode dite de Spectral Count permet de mesurer de manière semi-quantitative l’abondance relative des protéines d’un échantillon à partir du nombre de fois où les peptides d’une protéine ont été analysés (ou fragmentés) par le spectromètre de masse.
Références :
AT_CHLORO: A comprehensive chloroplast proteome database with subplastidial localization and curated information on envelope proteins. Ferro M, Brugière S, Salvi D, Seigneurin-Berny D, Court M, Moyet L, Ramus C, Miras S, Mellal M, Le Gall S, Kieffer-Jaquinod S, Bruley C, Garin J, Joyard J, Masselon C, Rolland N. Mol. Cell. Proteomics, online, 2010
Ce projet a été réalisé dans le cadre du programme Génoplante (ANR), et la base de données est hébergée par l’INRIA (serveur PRABIG).
Source: communiqué de presse du CNRS
Laisser un commentaire