Une collaboration euro-asiatique de scientifiques impliquant tout particulièrement le CNRS, l’Institut Pasteur à Paris, l’Institut Pasteur de Corée à Séoul (IP Korea/Equipe Avenir Inserm (1)) et l’Université de Toulouse vient d’identifier dix gènes de virulence du bacille de la tuberculose. L’inactivation de ces gènes atténue le pouvoir pathogène du bacille. Cette découverte, publiée dans la revue PLoS Pathogens, permettra notamment de proposer des stratégies thérapeutiques inédites et de tester de nouveaux candidats-vaccins contre la tuberculose. Pour parvenir à de tels résultats en seulement deux semaines, les chercheurs ont d’abord mis au point une nouvelle technique de criblage. Cette méthode innovante, rapide et performante pourrait être facilement transposable à d’autres pathogènes intracellulaires.
Malgré les médicaments existants et la vaccination par le BCG, la tuberculose continue de faire des ravages : elle tue chaque année près de 2 millions de personnes dans le monde. Cette maladie est causée par des bactéries de la famille des mycobactéries, parmi lesquelles figure Mycobacterium tuberculosis, l’agent de la tuberculose chez l’homme. Sa virulence, c’est-à-dire sa pathogénicité, dépend de sa capacité à se propager au sein de la cellule hôte. Cet agent pathogène est en effet capable d’échapper aux défenses de l’hôte qu’il infecte en parasitant les macrophages, cellules du système immunitaire normalement impliquées dans l’ingestion et la destruction des microbes. Après inhalation, Mycobacterium tuberculosis se retrouve dans les poumons. Là, il est ingéré par les macrophages alvéolaires et réside dans un compartiment intracellulaire appelé « phagosome ». D’ordinaire, ce dernier a pour fonction de détruire, en s’acidifiant, les corps ingérés par le macrophage. Mais, au lieu d’être tué par la cellule, le bacille tuberculeux s’y multiplie en bloquant l’acidification du phagosome. Les gènes du bacille impliqués dans ce processus restaient jusqu’à présent méconnus. Les identifier fut l’un des objectifs de la collaboration euro-asiatique pilotée par Olivier Neyrolles, chercheur CNRS à l’Institut de pharmacologie et de biologie structurale (CNRS / Université de Toulouse) et Priscille Brodin, chercheuse à l’Inserm (Institut Pasteur de Corée / Inserm).
Pour cela, les chercheurs ont tout d’abord conçu une nouvelle méthode de criblage basée sur la sélection, par un robot, d’un phénotype cellulaire caractérisé par un cliché de microscopie. Grâce à cette technique novatrice, les échantillons correspondant au critère souhaité sont repérés visuellement et automatiquement. Ce dispositif offre donc un gain de temps considérable pour identifier des composants microbiens impliqués dans le parasitisme des cellules.
Cette méthode originale et performante a été ensuite appliquée à une souche particulièrement virulente (2) de Mycobacterium tuberculosis. Elle a permis de passer au crible plus de 11000 mutants (3) du bacille tuberculeux, cela en à peine quelques semaines. Le robot était alors programmé pour détecter si la fonction « acidification » était active ou non. Les chercheurs sont ainsi parvenus à isoler les mutants qui ne parvenaient pas à bloquer l’acidification du phagosome et qui étaient donc détruits par les macrophages. Les mutations correspondantes ont ensuite été identifiées par génie génétique, puis dix gènes, dans leur majorité inconnus, impliqués dans le parasitisme du macrophage ont été caractérisés.
La plupart de ces gènes codent pour la synthèse de produits sécrétés par la bactérie : protéines et lipides, dont la fonction précise dans le parasitisme des cellules par Mycobacterium tuberculosis reste désormais à élucider. De plus, ces molécules pourraient constituer des cibles de choix pour de nouveaux antibiotiques. Enfin, ces résultats suggèrent que les mutants isolés, dont certains sont atténués dans leur virulence in vivo, pourraient être de bons candidats pour la conception de nouveaux vaccins à même de remplacer le BCG.
Ces travaux ont bénéficié de plusieurs soutiens financiers, en particulier de l’Union Européenne dans le cadre du programme TB-VIR, coordonné par Olivier Neyrolles.
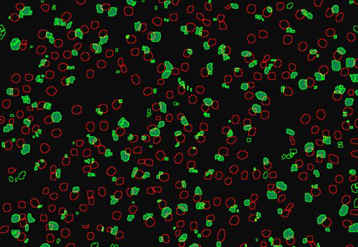
La méthode de criblage automatique permet d'identifier les cellules et les bactéries comme des "objets visuels" selon leur contour : les noyaux cellulaires apparaissent en rouge, les bactéries résidant dans un « phagosome » acidifié apparaissent en vert. L'automate et l'ordinateur quantifient ensuite le nombre et la surface des "objets" rouges et verts, permettant ainsi le criblage. © Institut Pasteur de Corée
Notes :
(1) Le programme ATIP-Avenir mené conjointement par l’Inserm et le CNRS donne les moyens à de jeunes chercheurs de haut niveau de mettre en place et d’animer une équipe de recherche pendant 3 ans. Depuis leurs créations, ces deux programmes ont permis à plus de 438 chercheurs de constituer leur propre équipe de recherche dans les domaines des sciences de la vie et de la santé.
(2) Il s’agit de la famille appelée « Beijing » pour sa prépondérance en Asie. Elle est aujourd’hui considérée comme la seconde par sa fréquence mondiale et est fréquemment associée à une pharmacorésistance.
(3) Le génome du bacille tuberculeux comporte plus de 4000 gènes. Les chercheurs ne connaissent pas encore la fonction de la moitié de ces gènes environ. Cela met en évidence la pertinence de méthodes de criblage à haut débit : avec une banque de 11 000 mutants pour un génome comportant 4000 gènes, les chercheurs sont certains d’avoir couvert l’ensemble du génome du bacille.
Références :
High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. Priscille Brodin, Yannick Poquet, Florence Levillain, Isabelle Peguillet, Fanny Ewann, Gerald Larrouy-Maumus, Martine Gilleron, Thierry Christophe, Denis Fenistein, Jichan Jang, Mi-Seon Jang, Park Sei-Jin, Jean Rauzier, Jean-Philippe Carralot, Rachel Shrimpton, Auguste Genovesio, Jesus A. Gonzalo Asensio, Germain Puzo, Carlos Martin, Roland Brosch, Graham R. Stewart, Brigitte Gicquel and Olivier Neyrolles. PLoS Pathogens, 9 septembre 2010
Source: communiqué de presse du CNRS
Laisser un commentaire