Jusqu’à présent, la microscopie de super-résolution, également appelée nanoscopie, autorisait la cartographie d’une population de molécules individuelles à la surface de cellules vivantes, à condition de modifier génétiquement les organismes pour les rendre fluorescents. Des physiciens et biologistes du CNRS et de l’Université de Bordeaux ont étendu cette technique à l’imagerie de molécules se trouvant à la surface de cellules non modifiées génétiquement et au suivi de leurs trajectoires pendant plusieurs secondes. Cette nouvelle technique permet un grand nombre d’études jusqu’à présent inaccessibles et suscite un énorme intérêt dans la communauté des biologistes cellulaires.
Le microscope est l’outil idéal pour observer le monde vivant à petite échelle. Quel élève n’a pas observé les cellules des pelures d’oignons ou les chloroplastes entre deux petites plaquettes de verre ? Mais lorsque l’on s’intéresse à des objets encore plus petits à l’intérieur des cellules, le phénomène de diffraction (1) limite la résolution : en deçà de 250 nanomètres (un quart de micron), les objets que l’on veut observer ne sont plus que des taches, car les pinceaux lumineux renvoyés par chaque point de l’objet s’élargissent et se confondent.
La détection de molécules uniques
Depuis quelques années, les chercheurs ont réussi à contourner cette difficulté grâce une famille de nouvelles techniques de « microscopies de super-résolution » ou nanoscopies. Ces méthodes, capables de fournir des images nanoscopiques des assemblages moléculaires présents à la surface des cellules, sont en passe de révolutionner le domaine de l’imagerie cellulaire biologique. Quel est leur principe ? On utilise le fait que malgré la diffraction, on peut localiser très précisément un point (ou une molécule) unique, puisque l’on sait qu’il se trouve au centre de la tache de lumière qu’il nous renvoie, alors que dès que l’on a plusieurs points sur un objet dont on veut faire une image, les deux tâches se recouvrent et on ne peut plus connaître leurs positions exactes.
Les chercheurs ont eu l’idée d’utiliser l’ingénierie génétique pour n’ « allumer », à un instant donné, que quelques points de façon aléatoire sur les objets qu’ils souhaitaient imager à la surface de cellules vivantes. Un peu comme une guirlande de Noël où les ampoules s’allument les unes après les autres et qui permet au final d’avoir une idée de la forme globale du sapin, une fois que toutes les ampoules se sont allumées.
Mais ces méthodes présentent un inconvénient majeur : il faut être capable d’allumer et d’éteindre les molécules une à une, à la demande. Pour cela, on utilise des protéines fluorescentes particulières introduites dans les cellules par ingénierie génétique. Deux lasers permettent de n’allumer statistiquement que quelques molécules à la fois. Ces molécules fluorescentes constituent quelques points de l’image. L’opération est répétée pour obtenir les autres points et former, au final, l’image complète de l’assemblage moléculaire à étudier. Contraints d’utiliser des protéines fluorescentes particulières, les chercheurs ne pouvait pas étudier de molécules biologiques à la surface des cellules sans les modifier génétiquement, ni suivre leur trajectoire pendant plusieurs secondes. En effet, les propriétés de fluorescence des molécules modifiées par génie génétique durent pour la plupart bien moins d’une seconde.
Aujourd’hui, les physiciens du Centre de physique moléculaire optique et hertzienne et les biologistes du laboratoire Physiologie cellulaire de la synapse (deux laboratoires CNRS/Université de Bordeaux) ont développé une nouvelle technique pour « allumer » les molécules : ils ont utilisé l’immuno-marquage en temps réel. Les cellules vivantes sont placées en présence d’une solution d’anticorps fluorescents (disponibles dans le commerce, un anticorps correspondant à chaque type d’objet étudié), qui vont se lier à elles. Mais au lieu de mettre suffisamment d’anticorps pour se lier à toutes les molécules à étudier, on réalise un marquage dilué sous le microscope : les quelques anticorps disponibles vont se lier aux molécules à étudier et les rendre lumineuses, puis, quand elles seront éteintes, d’autres seront déjà en train de se lier au nouveaux anticorps qui arrivent dans l’échantillon (le marquage se fait en continu) et s’allumeront à leur tour.
« Filmer » les cellules avec une résolution de 50 nanomètres
La technique permet d’enregistrer plusieurs dizaines de milliers de trajectoires de molécules uniques sur une seule cellule et d’en étudier la dynamique à l’échelle nanométrique. Les chercheurs ont démontré son efficacité sur divers systèmes cellulaires (cellules hétérologues, fibroblastes ou neurones en cultures) pour étudier différentes molécules membranaires (2). La simplicité, l’adaptabilité et la fiabilité de cette nouvelle méthode permettent d’ores et déjà d’envisager un grand nombre d’études inaccessibles jusqu’alors par la microscopie optique conventionnelle. En particulier, il devient possible de « filmer » l’évolution d’une seule cellule, par exemple un neurone, avec une résolution spatiale de 50 nanomètres pour une cadence d’imagerie correspondant à celle de la vidéo (20 images pas seconde). Les chercheurs bordelais ont déjà entamé l’étude de la structuration dynamique des récepteurs de neurotransmetteurs dans les synapses de neurones vivants. Nul doute qu’au vu du très grand nombre d’équipes déjà intéressées par cette nouvelle technique, elle devrait déclencher une petite révolution dans le monde de l’imagerie biologique.
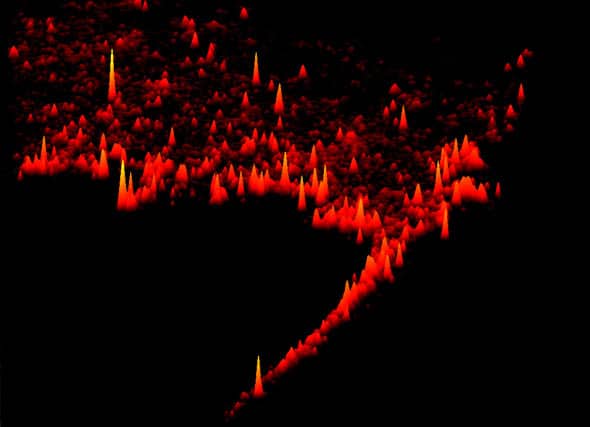
Image super-résolue d'une protéine transmembranaire à la surface d'un fibroblaste : les pics les plus hauts correspondant à 70 détections de ces complexes dans 50x50nm², (l'image fait environ 10 microns de large). La technique permet d'acquérir 20 images par secondes, ce qui permet de reconstituer les trajectoires. © © Gregory. Giannone, Eric. Hosy, Florian Levet.
Notes :
(1) On peut comprendre ce qu’est la diffraction lorsque l’on place une feuille percée d’un petit trou devant une lampe et que l’on observe sur un écran la tache lumineuse formée. Si le trou est suffisamment grand, la tâche a la même dimension que le trou. Lorsqu’il atteint des dimensions de l’ordre de la longueur d’onde de la lumière visible (de 400 à 700 nanomètres), plus le trou est petit, plus la tache est étendue en comparaison. Il n’est donc pas possible d’avoir un pinceau de lumière aussi fin qu’on le souhaite.
(2) en collaboration avec un informaticien de l’INSERM, des chimistes de Francfort (Allemagne) et des biologistes de Portland (USA).
Références :
Dynamic super-resolution imaging of endogenous proteins on living cells at ultra-high density, G. Giannone, E. Hosy, F. Levet, A. Constals, K. Schulze, A.I. Sobolevsky, M.P. Rosconi, E. Gouaux, R. Tampé, D. Choquet and L. Cognet, Biophysical Journal, 18 août 2010.
Source: communiqué de presse du CNRS
Laisser un commentaire